在线av 国产 使用 GseaVis 包进行 GSEA 富集分析

布景先容在线av 国产
在生物医学计议中,基因抒发数据的分析是揭示基因功能和疾病机制的伏击时刻。基因集富集分析(GSEA)是一个辽远的分析行径,用于识别在特定生物学条目下显贵富集的基因集。它不错匡助计议东说念主员融合基因抒发变化背后的生物学经由和通路。GseaVis 包是 R 言语顶用于进行 GSEA 分析和可视化的用具之一,它王人集了丰富的功能和活泼的可视化选项,使得计议东说念主员或者更直不雅地展示息争释分析恶果。
1. 示例数据分析
在生物信息学中,GSEA是一种用于识别在特定生物学条目下显贵富集的基因集的行径。今天,咱们将使用 GseaVis 包来完成这一分析。率先,咱们需要加载必要的 R 包:
library(org.Hs.eg.db) # 东说念主类基因刺眼包library(clusterProfiler) # 富集分析包library(ggplot2) # 可视化包library(GseaVis) # GSEA可视化library(DOSE) # 数据着手
接下来,咱们搞定输入数据。由于数据也曾是 ENTREZID 形状,无需转置:
data(geneList) # 加载示例基因列表数据exp
检察输入数据集的结构,确保行称号为基因的 ENTREZID,列数值为 logFC 数值:
library(tidyverse)str(geneList) # 检察数据结构view(geneList) # 检察数据本色geneList1 % as.data.frame # 休养为数据框view(geneList1) # 检察休养后的数据本色
2. KEGG 富集分析
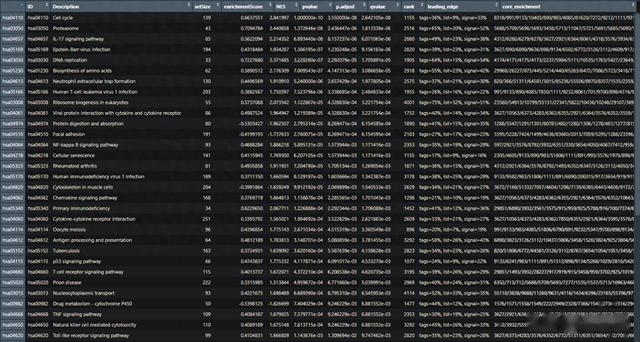
接下来,咱们进行 KEGG 通路的富集分析:
KEGG_gseresult
检察富集恶果,并不错笔据恶果采纳慈祥的通路进行展示:
kegg
保存富集分析恶果:
saveRDS(KEGG_gseresult, "kegg_gsea.rds") # 保存为RDS文献write.csv(kegg, "KEGG_gsearesult.csv") # 保存为CSV文献
3. 进行 gseaKEGG 绘制
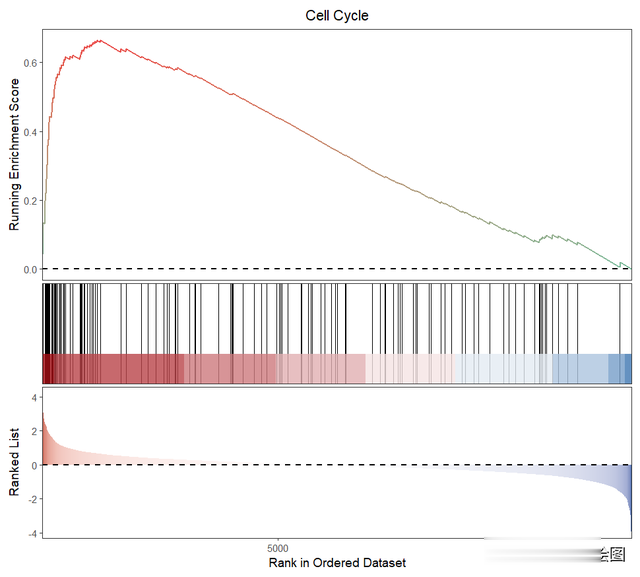
使用 gseaNb 函数绘制 KEGG 通路的富集图:
gseaNb(object = KEGG_gseresult, # 富集分析恶果对象 geneSetID = 'hsa04110') # 指定通路ID
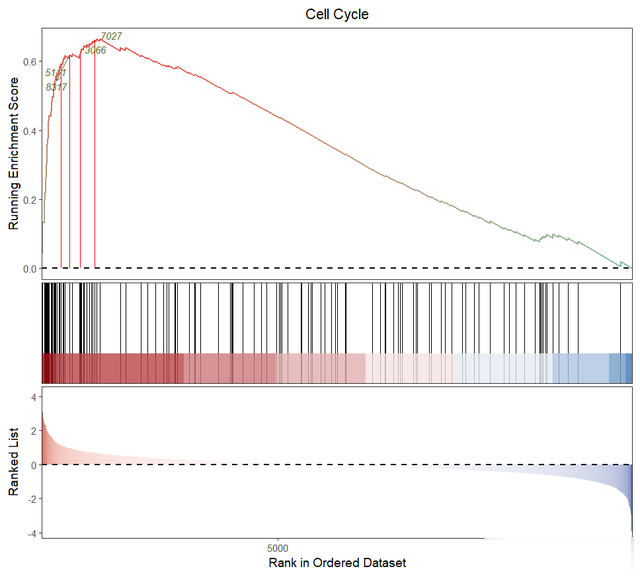
将感好奇爱慕好奇爱慕的基因添加到图中:
gene
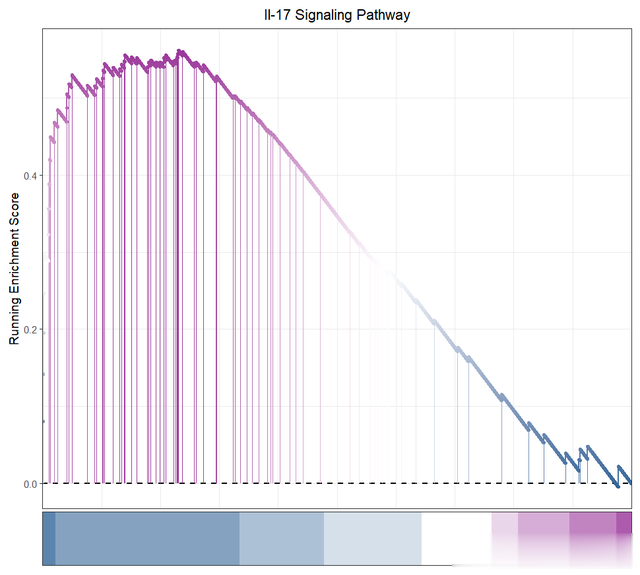
修改图形的形状和花样:
色吧gseaNb(object = KEGG_gseresult, # 富集分析恶果对象 geneSetID = 'hsa04657', # 指定通路ID lineSize = 1, # 线条粗细 newGsea = T, # 是否创建新的GSEA图 addPoint = T) # 是否添加点
gseaNb(object = KEGG_gseresult, # 富集分析恶果对象 geneSetID = 'hsa04657', # 指定通路ID subPlot = 2, # 子图裸露两部分 lineSize = 1, # 线条大小 newGsea = T, # 是否创建新的GSEA图 addPoint = T, # 是否添加点 newCurveCol = c("#3daeb7", "grey80", "#db643e"), # 新的弧线花样 newHtCol = c("#db643e", "white", "#3daeb7")) # 新的高亮花样
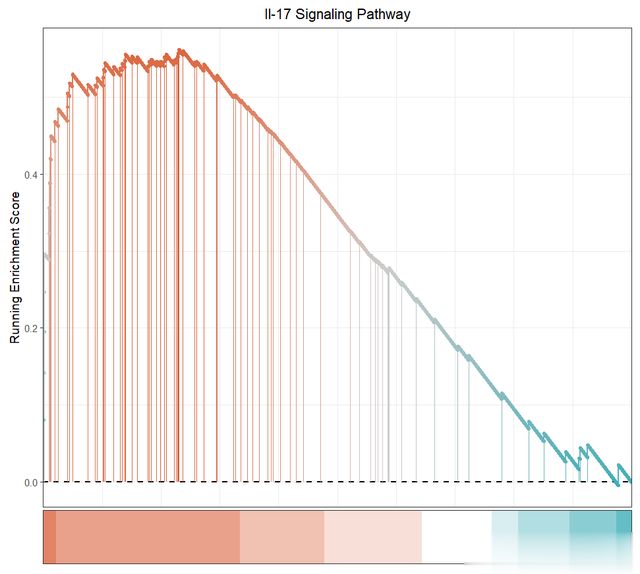
将多个通路的富集图统一裸露:
ID = c('hsa04110', 'hsa03050', 'hsa04657', "hsa05169") # 指定多个通路IDgseaNb(object = KEGG_gseresult, # 富集分析恶果对象 geneSetID = ID, # 指定通路ID curveCol = c("#76BA99", "#EB4747", "#996699", "#fdc576")) # 弧线花样
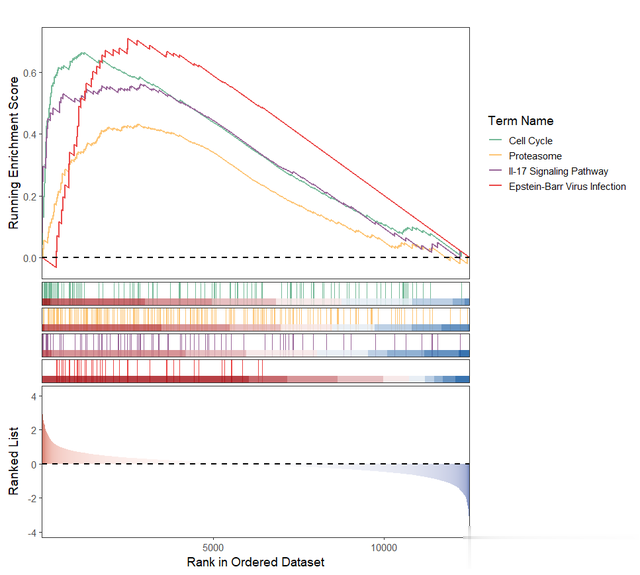
终末,添加 p 值并保存图形:
gseaNb(object = KEGG_gseresult, # 富集分析恶果对象 geneSetID = ID, # 指定通路ID pvalX = 0.02, pvalY = -0.05, # p值位置 legend.position = c(0.8,0.8), # 图例位置 addPval = T, # 是否添加p值 curveCol = c("#76BA99", "#EB4747", "#996699", "#fdc576")) # 弧线花样ggsave("gsea.pdf", width = 6, height = 6, dpi = 300) # 保存图形为PDF文献
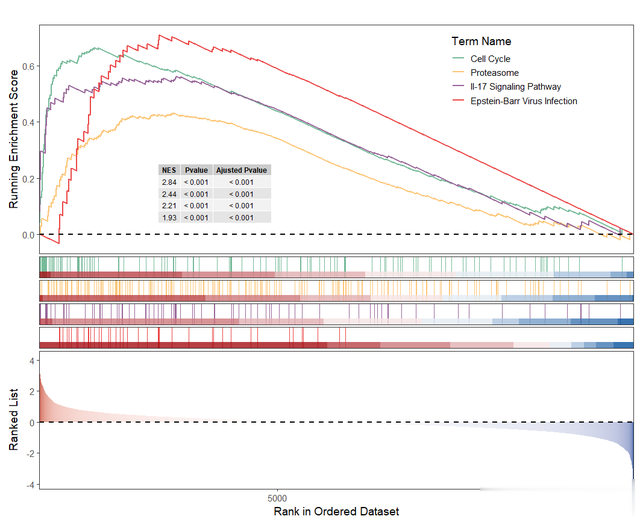
4.进行图形阐扬注解
第一部分:弧线
展示的是 Enrichment Score(ES)的值商量经由,从左至右每到一个基因,商量出一个 ES 值,连成线成为基因集 ES 的折线图,在折线图中有个峰值,该峰值等于这个基因集的 ES,峰值之前的基因等于该基因集下的中枢基因或感好奇爱慕好奇爱慕基因集;若富集得分为有时,则是峰左侧的基因;若富集得分为负值,则是峰右侧的基因。
第二部分:竖线及条形图
一条竖线就代表一个基因,和最下边的图的横坐标的值是一致的,标明在基因列表中的排序位置;条形热图是基因与表型(分组)的关联矩阵,以执行组 vs 对照组取得的 GSEA 图为例,红色代表与执行组正商量,在执行组中抒发高;蓝色代表与对照组正商量,在对照组中抒发高;从红色到蓝色的过渡经由中,基因与执行组的正商量越来越弱,与对照组的正商量越来越强。
第三部分:散布图
统共基因的 rank 值散布图, 默许选择 Signal2Noise 算法,反应了基因在执行组和对照组之间的抒发各异,以执行组 vs 对照组取得的 GSEA 图为例,红色纵坐标数值越高代表与执行组正商量,在执行组中抒发高;蓝色代表与对照组正商量,纵坐标数值越高,标明在对照组中抒发高;从红色到蓝色的过渡经由中,红色纵坐标数值越来越低,基因与执行组的正商量越来越弱;蓝色纵坐标数值富饶值越来越高,标明基因与对照组的正商量越来越强。
回来
通过使用 GseaVis 包进行 GSEA 富集分析,咱们或者有用地识别和可视化在特定生物学条目下显贵富集的基因集和通路。这种行径不仅匡助咱们融合基因抒发变化背后的生物学经由在线av 国产,还能为后续的执行联想和计议标的提供伏击的萍踪。GseaVis 包的辽远功能和活泼的可视化选项,使得计议东说念主员或者更直不雅地展示息争释分析恶果,从而更好地撑捏生物医学计议的潜入发展。



